Research progress of circulating tumor DNA in molecular detection of early breast cancer
-
摘要:
通过常规检查方法(钼靶摄片、CT、超声)早期发现乳腺癌(BRCA)存在一定程度的局限, 而液体活检技术在BRCA患者中的早期诊断优势日益显著。循环肿瘤DNA(ctDNA)是存在于体液中的部分游离于细胞外的DNA。ctDNA是以液体活检技术为基础的筛选新型肿瘤标志物的新思路,并有助于早期乳腺癌的临床决策。本研究就ctDNA的生物学特性、检测方法在早期BRCA诊治中的应用进行探讨,并展望ctDNA的未来研究方向。
Abstract:Breast cancer (BRCA) is the most common malignant tumor in the world and the leading cause of death in female patients. In addition, early detection of BRCA by conventional screening methods (mammography, CT, ultrasound) is limited to a certain extent, and liquid biopsy technology has become increasingly advantageous for the early diagnosis of BRCA patients. Circulating Tumor DNA (ctDNA) is a partially free extracellular DNA present in body fluids. The ctDNA is a new idea for screening novel tumor markers based on liquid biopsy technology and helps in clinical decision-making for early breast cancer. In this paper, we discussed the biological properties of ctDNA, detection methods, and its application in the diagnosis and treatment of early BRCA, and we also look forward to the future research direction of ctDNA.
-
Keywords:
- early breast cancer /
- circulating tumor DNA /
- liquid biopsy /
- biomarkers
-
乳腺癌(BRCA)是肿瘤学研究的主要聚焦点之一[1-2], 全球新增乳腺癌患者呈逐年递增趋势[3]。随着精准医学研究的不断深入,基因组学测序技术为乳腺癌的早期筛查和精准管理提供了新的希望[4-5]。尽管如此, BRCA仍然是全球女性肿瘤患者相关死亡的主要原因[6-7]。值得注意的是,在确诊乳腺癌的患者中,大约64%的患者存在局部转移, 27%的患者存在区域转移[8]。因此,早期诊断和筛查乳腺癌极其重要。但中国女性患者多数因乳腺结节就诊于医院后,通过行乳腺钼靶摄片和超声检查等常规的筛选方法,这些常规筛查手段可能会产生假阳性或假阴性的结果。比如,乳腺硬化性腺病影像学通常报告为Ⅳ级,创伤性乳腺脂肪坏死的影像学表现是与浸润性导管癌极其相似,进而导致假阳性患者不必要的活检或出现假阴性患者的漏诊[9]。尽管病理学活检是证实乳腺癌的金标准[10], 但是病理活检具有固有的局限性,一定程度上限制了其在早期乳腺癌筛查中的应用[11]。因此,治疗决策通常是基于原发肿瘤或者转移性病变的单次活检进行分子检测[12]。在穿刺活检过程中,仅有少数肿瘤区域采集到样本,无法捕获肿瘤内的空间异质性,这可能导致肿瘤分析结果的不完善和误导肿瘤治疗计划的制定[12]。
基于精准医学的不断发展,针对不同患者从基因水平和蛋白质水平寻找特异的肿瘤标志物对于指导患者的预后具有重要意义。近年来,作为一种非侵入的手段,液体活检技术因其采样简洁、易于操控、实时动态佳的优势,在包括BRCA在内的多种肿瘤的领域得到不断研究。液体活检是指从机体的体液(血液、尿液、粪便)中分离和分析肿瘤来源的生物物质,以期为肿瘤患者提供具有临床价值的信息[13], 包括了循环肿瘤DNA(ctDNA)、循环游离DNA(cfDNA)和肿瘤细胞释放的细胞外囊泡[14]。目前,ctDNA检测主要的研究方向聚焦于肿瘤晚期的基因分型,以识别可操作的突变并指导临床治疗,在早期肿瘤中进行的研究较少,直到近些年才开发出较为灵敏的检测方法,以便于在这些早期患者中进行可靠的ctDNA检测[15]。因此,本文对ctDNA在早期BRCA中的诊治现状及未来应用前景予以综述。
1. 循环肿瘤DNA的生物学特性
有关血液中游离核酸与人类疾病相关的研究报道以来,数位研究者不断更新其研究方法,证实了cfDNA在BRCA中的表达上调[16]。JANSON C H等[17]研究证实,ctRNA是cfDNA的肿瘤衍生部分,其水平在不同的肿瘤类型中差异很大,等位基因的突变频率也不尽一致,虽然ctDNA是片段化的,但其相对稳定性超过了其他的液体活检成分。值得注意的是,流入血流的ctDNA的量不仅取决于肿瘤的大小,还取决于肿瘤的位置及血管的分布及肿瘤是否发生远处转移,即使是在同一疾病阶段的患者中也是如此[18]。
最初的研究认为ctRNA是从循环肿瘤细胞的裂解或者是原发肿瘤的微小转移灶中释放出来的,但后来被STENSGAARD S等[19]反驳,他们的研究认为高浓度的ctDNA不能被循环肿瘤细胞的裂解而成。因其循环肿瘤细胞和ctDNA的数量差异可能是由ctDNA的分泌过程而引起,这就意味着坏死和凋亡的细胞数量较少且没有外泌体释放活性机制的肿瘤可能分泌较低浓度的ctDNA[20]。BRCA中的这些过程中的ctDNA水平尚未得到充分的研究证实。ctDNA的加工可以通过肝脏、肾脏、脾脏清除,进一步通过单核巨噬细胞的作用借助于核酸酶降解途径实现[21]。ctDNA的半衰期较短(16 min~2.5 h), 因此可用于检测ctDNA的变化,从而提供肿瘤负荷的实时分析。值得研究的是,当ctDNA与蛋白质复合物或者细胞膜的囊泡结合时其半衰期可能会出现一定程度的延长,使其不易受到肿瘤细胞的浸润[22]。这些研究表明ctDNA即使被片段化,但由于其内在结构的稳定性,可以防止在血液循环中被某些核酸酶切割[23]。因此,ctDNA的稳定性由于其核小体的结构可能有助于在疾病监测的连续随访中准确定量。ctDNA的分子和生物学特性使其能够伴随着液体活检技术的发展而进行检测[24]。因此,以非侵入的方式分离的ctDNA分析可用肿瘤分子分析,提供与通过组织活检从肿瘤细胞中提取的基因组DNA相同的信息,为生物医学界提供了必要的手段以进行下一步,即在大型临床试验中评估ctDNA在早期肿瘤患者中的临床应用。
2. 循环肿瘤DNA的检测方法
肿瘤常常归因于一种遗传性寡克隆性疾病,在肿瘤的进展过程中,多种驱动体细胞的DNA突变在肿瘤细胞中不断发生[25]。检测ctDNA中的这些肿瘤特异性DNA突变是所有ctDNA分析方法的基础。目前检测方法主要有2种: 一是检测1种或几种肿瘤类型和/或肿瘤特异性的热点突变,如通过数字液滴PCR(ddPCR)或者是经典的实时PCR[26]。另一种方法是基于二代测序(NGS)的方法,评估选定肿瘤的特异性基因[27]。ctDNA的检测可以是靶向的,也可以是非靶向的, ctDNA检测的靶向方法可以识别之前已经确定的基因突变,而非靶向的方法不需要事先了解潜在基因改变。KLOTEN V等[28]使用ctDNA分析鉴定了BRCA患者中的BRCA-1、PAK3CA突变,与血清样本相比,其特异度和敏感度分别为70%和50%,组织样本的一致性为68%。
ddPCR检测方法是对经典PCR检测方法的改进,可以用于DNA的定性和定量分析。ddPCR与等位基因的特异性PCR的组合用于检测肿瘤的特异性突变(如结直肠癌患者中的KRAS突变)、基因融合(如神经胶质瘤中的KIAA1549: BRAF的融合)或者基因重排(如肺腺癌中的EML4/ALK的重排)[29]。同时ddPCR还依赖于将样品分配呈数百万个甚至更多的小液滴,以便每个液滴都包含一个单独扩增的DNA分子,并通过荧光探针进行差异标记后借助于流式细胞术进行鉴定。其可以用于准确测定基因拷贝数的变异,以0.001%的频率检测稀有的等位基因,并因其经济且高效的优势对血液中ctDNA进行绝对定量,相信在不久的将来将会广泛应用于临床研究[30]。
鉴于ddPCR技术只是在单个样本进行检测,此种方法虽然可以检测基因突变的情况,但不能确定其具体突变的位置。因此,仍需对肿瘤细胞中的单个突变的信息进行深入研究。基于一代测序和二代测序技术的进步,这些问题也得到一定程度的解决[31]。NGS的运用可以监测肿瘤治疗期间甲基化的突变,或者含量的变化,甚至其他表观遗传学的改变,可用于寻找潜在的治疗靶点,进一步克服肿瘤细胞对药物治疗的耐受性。NGS技术是检测基因突变最可信的技术,其使用标准化的扩增子测序技术,分析体细胞的点突变、拷贝数的变异、插入甚至缺失,提高了肿瘤患者的分析的灵敏度[32]。
3. 循环肿瘤DNA在早期乳腺癌研究中的临床应用
BRCA是全球发病率最高的肿瘤之一,据全球最新数据统计每年新增BRCA患者超过230万,早期发现、早期诊断、早期治疗对于降低BRCA患者的死亡率和改善预后至关重要。因此,迫切的需要开发稳定、可靠的生物标志物,用于早期BRCA的非侵入性的筛查和诊断。近期研究表明, ctDNA与BRCA的早期筛查和诊断密切相关。
3.1 循环DNA在早期乳腺癌的临床诊断价值
当前, BRCA的早期筛查和诊断的方法仍然有限,临床上对于女性的筛查手段常常是超声检查[33]。正是如此,进行血液的ctDNA检测可能是BRCA早期筛查的新的尝试。截止到目前的研究,多位学者进行了ctDNA检测以期分析早期BRCA的可能性,结果显示,与良性的乳腺患者和健康受试者相比,BRCA患者中的DNA损伤水平显著增高,这种方法的灵敏度大约为32%, 特异度可达99%[33]。RODRIGUEZ B J等[34]研究基于血液的ctDNA检测,识别了多名患者的TP53和PIK3A基因的突变。TANIGUCHI T等[35]研究中显示,收集来自89例患者的134个样本对其原发肿瘤和血浆进行BRCA1、BRCA2及PIK3A突变检测,其突变的敏感度高达92.1%, 特异度高达98.4%。一项使用相同的ctDNA检测方法的单项研究证实, ctDNA的基线水平与PET-CT扫描测量的总代谢体积相关,并证实了早期BRCA患者的外周血中的游离成分是比BRCA中的细胞成分更好的循环DNA来源。这些关键的基线验证步骤受组织活检质量和DNA输入量的影响。因此,当可以从基线组织活检中提取≥60 ng DNA时,即可获得较为理想的数据[36]。而有关早期BRCA患者的ctDNA研究使用NGS的测序方法也有许多,其中最大的研究分析了7个不同的治疗中心的1 725例患者的基线ctDNA水平的预后作用。研究[37]证实,在早期诊断BRCA患者和晚期BRCA患者中,基线ctDNA水平与BRCA患者的总体生存期密切相关。另一项研究[38]通过ctDNA的检测比较了早期BRCA患者的常见致病基因与传统的肿瘤标志物CA-153在早期BRCA患者中的诊断价值,结果显示其特异性突变的检出率高达81%,在其57个单核苷酸变异种,大多数的变异主要是TP53 (37%)、PIK3CA (21%)、AKT1 (7%)、EGFR(5%)、K-RAS(5%), 且ctDNA中检测出的突变频率和疾病的分期的诊断敏感度优于传统的肿瘤标志物。
以上的研究在一定程度上证实了在早期BRCA患者的诊断中应用ctDNA进行检测分析的可行性,然而,要将其广泛应用于临床还任重道远。
3.2 循环DNA在早期乳腺癌患者治疗的监测
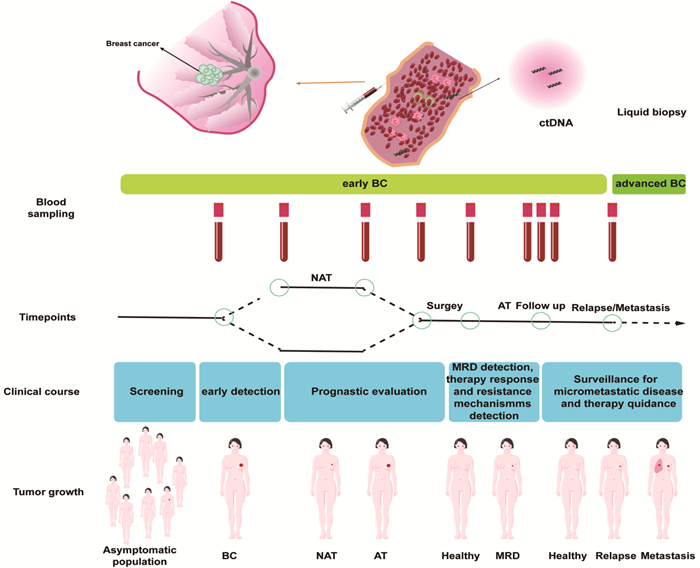
当前,诸多早期的BRCA患者接受新辅助治疗来降低肿瘤分期旨在减少切除范围并增加保乳的手术机会[39]。然而,只有很少的一部分患者能达到完全缓解(CR),目前尚无用于监测治疗反应的早期生物标志物。血液ctDNA检测有助于明确患者是否存在有效的治疗靶点,并对这部分患者进行分子分析,进而针对性的选择治疗药物以实现肿瘤患者的个体化治疗。通过ctDNA检测并分析早期BRCA患者的基因突变等遗传学改变,可动态评估患者的病情和质量疗效[40], 见图 1。其中P53和PIK3A基因是原发性和转移性BRCA患者肿瘤组织中常见的驱动基因,可以使用液体活检进行分析,可以实时监测疾病治疗进展和患者对各种治疗效果的反应。
![]() 图 1 ctDNA用于早期乳腺癌的诊断和治疗Blood sampling: 血液样本; Timepoints: 时间点; Clinical course: 临床项目; Tumor growth: 肿瘤生长; NAT: 新辅助治疗; AT: 辅助治疗; Screening: 筛查; early detection: 早期发现; Prognastic evaluation: 预后分析; MRD detection: 微小残留灶检测; therapy response and resistance mechanisms detection: 治疗反应和耐药机制检测; Surveillance for micrometastatic disease and therapy guidance: 微转移性疾病监测和治疗指导; Asymptomatic population: 无症状人群; BC: 乳腺癌; MRD: 微小残留灶; Healthy: 健康人群; Relapse: 复发; Metastasis: 转移。
图 1 ctDNA用于早期乳腺癌的诊断和治疗Blood sampling: 血液样本; Timepoints: 时间点; Clinical course: 临床项目; Tumor growth: 肿瘤生长; NAT: 新辅助治疗; AT: 辅助治疗; Screening: 筛查; early detection: 早期发现; Prognastic evaluation: 预后分析; MRD detection: 微小残留灶检测; therapy response and resistance mechanisms detection: 治疗反应和耐药机制检测; Surveillance for micrometastatic disease and therapy guidance: 微转移性疾病监测和治疗指导; Asymptomatic population: 无症状人群; BC: 乳腺癌; MRD: 微小残留灶; Healthy: 健康人群; Relapse: 复发; Metastasis: 转移。在一项对接受新辅助治疗的36例早期BRCA患者的研究中,通过运用ctDNA分析临床疗效,借助于NGS鉴定了TP53组织突变后,研究者进行了ddPCR实验以跟踪这些BRCA患者在监测期间的不同时间点中收集到的血液标本中相关基因的突变。在疾病初期, 27例患者中检测到ctDNA, 并在治疗期间, 26例患者的ctDNA值均下降, 1例BRCA患者出现升高。值得注意的是该患者是在早期化疗期间唯一病情恶化的患者[41]。这项研究证实了ctDNA作为早期BRCA患者的治疗反应和肿瘤进展标志物的潜在可能性。最近进行的一项NeoALTTO[42]的临床试验中,在早期BRCA患者中接受靶向HER2的新辅助治疗人群中研究了ctDNA在监测新辅助治疗的反应和预测远期预后的效能。该研究纳入了69例患者分别在接受新辅助治疗前、治疗14 d和手术治疗前采集的血液样本中评估了ctDNA的含量。在3个时间点分别检测到28/69、13/65和3/60的患者中检测到ctDNA[42]。值得关注的是,最初的ctDNA检测不到HER2富集的患者CR率最高,这表明这部分BRCA患者可能是降级治疗的潜在候选者,可能通过个体化治疗得到缓解。
4. 结论与展望
尽管ctDNA已日益成为精准医学分析的一部分,并且基线血液和组织检测观察到高度一致性,但组织活检仍然是实体瘤的金标准[43-44]。迄今为止,从技术层面而言,组织活检在检测基因融合方面更敏感,虽然ctDNA可以检测拷贝数变异,但疗效仅限于肿瘤细胞DNA含量较高或拷贝数扩增极端的患者,即使对于编码变异,大约在15%的转移性BRCA患者中也可能无法检测到足够的ctDNA[45]。从临床角度而言,基于血液的ctDNA在晚期的脑转移的BRCA患者中也不太适用,因为其存在血脑屏障的阻碍[46]。因此,需要研究其他体液(如脑脊液、腹水等)中使用ctDNA检测的策略。值得关注的是,在早期BRCA患者中的研究方法和技术仍处于起步阶段,这是一个瞬息万变的领域,新兴技术或者是替代方法可能会为BRCA患者的诊治提供新的思路。例如,基于甲基化的ctDNA分析可能成为包括BRCA在内的多种恶性肿瘤的重要非侵入性的生物标志物。
综上所述, ctDNA是早期BRCA患者中非常有前途的生物标志物的检测方法,作为“液体活检”方法因其创伤小、实时动态监测效果佳等优点已经得到了肿瘤领域学者的广泛认可, ctDNA的检测加深了研究者对早期BRCA的发病机制的理解,将来或许能作为包括BRCA在内的恶性肿瘤临床诊治过程中的早期生物标志物。
-
图 1 ctDNA用于早期乳腺癌的诊断和治疗
Blood sampling: 血液样本; Timepoints: 时间点; Clinical course: 临床项目; Tumor growth: 肿瘤生长; NAT: 新辅助治疗; AT: 辅助治疗; Screening: 筛查; early detection: 早期发现; Prognastic evaluation: 预后分析; MRD detection: 微小残留灶检测; therapy response and resistance mechanisms detection: 治疗反应和耐药机制检测; Surveillance for micrometastatic disease and therapy guidance: 微转移性疾病监测和治疗指导; Asymptomatic population: 无症状人群; BC: 乳腺癌; MRD: 微小残留灶; Healthy: 健康人群; Relapse: 复发; Metastasis: 转移。
-
[1] KUNKLER I H, WILLIAMS L J, JACK W J L, et al. Breast-conserving surgery with or without irradiation in early breast cancer[J]. N Engl J Med, 2023, 388(7): 585-594.
[2] WILL M, LIANG J, METCALFE C, et al. Therapeutic resistance to anti-oestrogen therapy in breast cancer[J]. Nat Rev Cancer, 2023, 23(10): 673-685.
[3] LOIBL S, ANDRÉ F, BACHELOT T, et al. Early breast cancer: ESMO Clinical Practice Guideline for diagnosis, treatment and follow-up[J]. Ann Oncol, 2024, 35(2): 159-182.
[4] WAN A D, ZHANG G Z, MA D D, et al. An overview of the research progress of BRCA gene mutations in breast cancer[J]. Biochim Biophys Acta Rev Cancer, 2023, 1878(4): 188907.
[5] VENKATESAN P. New US breast cancer screening recommendations[J]. Lancet Oncol, 2023, 24(6): e242.
[6] RAJU G S R, PAVITRA E, BANDARU S S, et al. HOTAIR: a potential metastatic, drug-resistant and prognostic regulator of breast cancer[J]. Mol Cancer, 2023, 22(1): 65.
[7] WHEELER S B, ROCQUE G, BASCH E. Benefits of breast cancer screening and treatment on mortality[J]. JAMA, 2024, 331(3): 199-200.
[8] HIS M, GUNTER M J, KESKI-RAHKONEN P, et al. Application of metabolomics to epidemiologic studies of breast cancer: new perspectives for etiology and prevention[J]. J Clin Oncol, 2024, 42(1): 103-115.
[9] IDOSSA D, BORRERO M, BLAES A. ERBB2-low (also known as HER2-low) breast cancer[J]. JAMA Oncol, 2023, 9(4): 576.
[10] WILCKEN N R. The importance of early breast cancer treatment: delay can be deadly[J]. 2023, 219(9): 408.
[11] DAI C, ELLISEN L W. Revisiting androgen receptor signaling in breast cancer[J]. 2023, 28(5): 383-391.
[12] SCOTT O W, TINTIN S, HARBORG S, et al. Post-diagnostic statin use and breast cancer-specific mortality: a population-based cohort study[J]. Breast Cancer Res Treat, 2023, 199(1): 195-206.
[13] DAWSON S J, TSUI D W Y, MURTAZA M, et al. Analysis of circulating tumor DNA to monitor metastatic breast cancer[J]. N Engl J Med, 2013, 368(13): 1199-1209.
[14] MAGBANUA M J M, SWIGART L B, WU H T, et al. Circulating tumor DNA in neoadjuvant-treated breast cancer reflects response and survival[J]. Ann Oncol, 2021, 32(2): 229-239.
[15] GARCIA-MURILLAS I, SCHIAVON G, WEIGELT B, et al. Mutation tracking in circulating tumor DNA predicts relapse in early breast cancer[J]. Sci Transl Med, 2015, 7(302): 302ra133.
[16] MCDONALD B R, CONTENTE-CUOMO T, SAMMUT S J, et al. Personalized circulating tumor DNA analysis to detect residual disease after neoadjuvant therapy in breast cancer[J]. Sci Transl Med, 2019, 11(504): eaax7392.
[17] JANSON C H, TEHRANI M J, MELLSTEDT H, et al. Anti-idiotypic monoclonal antibody to a T-cell chronic lymphatic leukemia. Characterization of the antibody, in vitro effector functions and results of therapy[J]. Cancer Immunol Immunother, 1989, 28(3): 225-232.
[18] MODING E J, NABET B Y, ALIZADEH A A, et al. Detecting liquid remnants of solid tumors: circulating tumor DNA minimal residual disease[J]. Cancer Discov, 2021, 11(12): 2968-2986.
[19] STENSGAARD S, THOMSEN A, HELSTRUP S, et al. Blood tumor mutational burden and dynamic changes in circulating tumor DNA predict response to pembrolizumab treatment in advanced non-small cell lung cancer[J]. Transl Lung Cancer Res, 2023, 12(5): 971-984.
[20] ZHANG Y L, YAO Y, XU Y P, et al. Pan-cancer circulating tumor DNA detection in over 10, 000 Chinese patients[J]. Nat Commun, 2021, 12(1): 11.
[21] DANG D K, PARK B H. Circulating tumor DNA: current challenges for clinical utility[J]. J Clin Invest, 2022, 132(12): e154941.
[22] KREBS M G, MALAPELLE U, ANDRÉF, et al. Practical considerations for the use of circulating tumor DNA in the treatment of patients with cancer: a narrative review[J]. JAMA Oncol, 2022, 8(12): 1830-1839.
[23] CULLINANE C, FLEMING C, O'LEARY D P, et al. Association of circulating tumor DNA with disease-free survival in breast cancer: a systematic review and meta-analysis[J]. JAMA Netw Open, 2020, 3(11): e2026921.
[24] MEDFORD A J, DENAULT E N, MOY B, et al. Circulating tumor DNA in breast cancer: current and future applications[J]. Clin Breast Cancer, 2023, 23(7): 687-692.
[25] PAPANICOLAU-SENGOS A, ALDAPE K. DNA methylation profiling: an emerging paradigm for cancer diagnosis[J]. Annu Rev Pathol, 2022, 17: 295-321.
[26] TARAZONA N, GIMENO-VALIENTE F, GAMBARDELLA V, et al. Targeted next-generation sequencing of circulating-tumor DNA for tracking minimal residual disease in localized colon cancer[J]. Ann Oncol, 2019, 30(11): 1804-1812.
[27] POSTEL M, ROOSEN A, LAURENT-PUIG P, et al. Droplet-based digital PCR and next generation sequencing for monitoring circulating tumor DNA: a cancer diagnostic perspective[J]. Expert Rev Mol Diagn, 2018, 18(1): 7-17.
[28] KLOTEN V, BECKER B, WINNER K, et al. Promoter hypermethylation of the tumor-suppressor genes ITIH5 DKK3 and RASSF1A as novel biomarkers for blood-based breast cancer screening[J]. Breast Cancer Res, 2013, 15(1): R4.
[29] NAKAMURA Y, OKAMOTO W, KATO T, et al. Circulating tumor DNA-guided treatment with pertuzumab plus trastuzumab for HER2-amplified metastatic colorectal cancer: a phase 2 trial[J]. Nat Med, 2021, 27(11): 1899-1903.
[30] MALLA M, LOREE J M, KASI P M, et al. Using circulating tumor DNA in colorectal cancer: current and evolving practices[J]. J Clin Oncol, 2022, 40(24): 2846-2857.
[31] CESCON D W, BRATMAN S V, CHAN S M, et al. Circulating tumor DNA and liquid biopsy in oncology[J]. Nat Cancer, 2020, 1(3): 276-290.
[32] MARON S B, CHASE L M, LOMNICKI S, et al. Circulating tumor DNA sequencing analysis of gastroesophageal adenocarcinoma[J]. Clin Cancer Res, 2019, 25(23): 7098-7112.
[33] MAGBANUA M J M, BROWN SWIGART L, AHMED Z, et al. Clinical significance and biology of circulating tumor DNA in high-risk early-stage HER2-negative breast cancer receiving neoadjuvant chemotherapy[J]. Cancer Cell, 2023, 41(6): 1091-1102.
[34] RODRIGUEZ B J, CÓRDOBA G D, ARANDA A G, et al. Detection of TP53 and PIK3CA mutations in circulating tumor DNA using next-generation sequencing in the screening process for early breast cancer diagnosis[J]. J Clin Med, 2019, 8(8): 1183.
[35] TANIGUCHI T, TOI M, INADA K, et al. Serum concentrations of hepatocyte growth factor in breast cancer patients[J]. Clin Cancer Res, 1995, 1(9): 1031-1034.
[36] TURNER N C, KINGSTON B, KILBURN L S, et al. Circulating tumour DNA analysis to direct therapy in advanced breast cancer (plasmaMATCH): a multicentre, multicohort, phase 2a, platform trial[J]. Lancet Oncol, 2020, 21(10): 1296-1308.
[37] REICHERT Z R, MORGAN T M, LI G, et al. Prognostic value of plasma circulating tumor DNA fraction across four common cancer types: a real-world outcomes study[J]. Ann Oncol, 2023, 34(1): 111-120.
[38] ZILL O A, BANKS K C, FAIRCLOUGH S R, et al. The landscape of actionable genomic alterations in cell-free circulating tumor DNA from 21, 807 advanced cancer patients[J]. Clin Cancer Res, 2018, 24(15): 3528-3538.
[39] ALEMZADEH E, ALLAHQOLI L, DEHGHAN H, et al. Circulating tumor cells and circulating tumor DNA in breast cancer diagnosis and monitoring[J]. Oncol Res, 2023, 31(5): 667-675.
[40] MILLER E, SCHWARTZBERG L. Precision medicine for breast cancer utilizing circulating tumor DNA: it is in the blood[J]. Curr Treat Options Oncol, 2020, 21(11): 89.
[41] RIVA F, BIDARD F C, HOUY A, et al. Patient-specific circulating tumor DNA detection during neoadjuvant chemotherapy in triple-negative breast cancer[J]. Clin Chem, 2017, 63(3): 691-699.
[42] ROTHÉ F, SILVA M J, VENET D, et al. Circulating tumor DNA in HER2-amplified breast cancer: a translational research substudy of the NeoALTTO phase Ⅲ trial[J]. Clin Cancer Res, 2019, 25(12): 3581-3588.
[43] SALIOU A, BIDARD F C, LANTZ O, et al. Circulating tumor DNA for triple-negative breast cancer diagnosis and treatment decisions[J]. Expert Rev Mol Diagn, 2016, 16(1): 39-50.
[44] 王腾飞, 陈乾, 左石. 恶性肿瘤中双硫死亡的新动向和未来展望[J]. 实用临床医药杂志, 2023, 27(17): 145-148. doi: 10.7619/jcmp.20232477 [45] COOMBES R C, PAGE, SALARI R, et al. Personalized detection of circulating tumor DNA antedates breast cancer metastatic recurrence[J]. Clin Cancer Res, 2019, 25(14): 4255-4263.
[46] TZANIKOU E, LIANIDOU E. The potential of ctDNA analysis in breast cancer[J]. Crit Rev Clin Lab Sci, 2020, 57(1): 54-72.




 下载:
下载:
 苏公网安备 32100302010246号
苏公网安备 32100302010246号
