Construction and validation of prognostic risk model for patients with hepatocellular carcinoma based on bioinformatics analysis
-
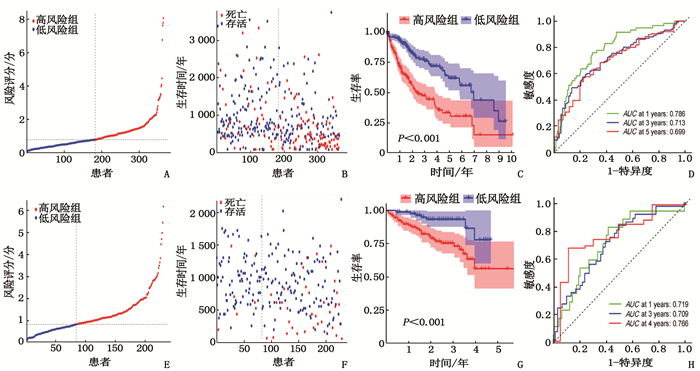
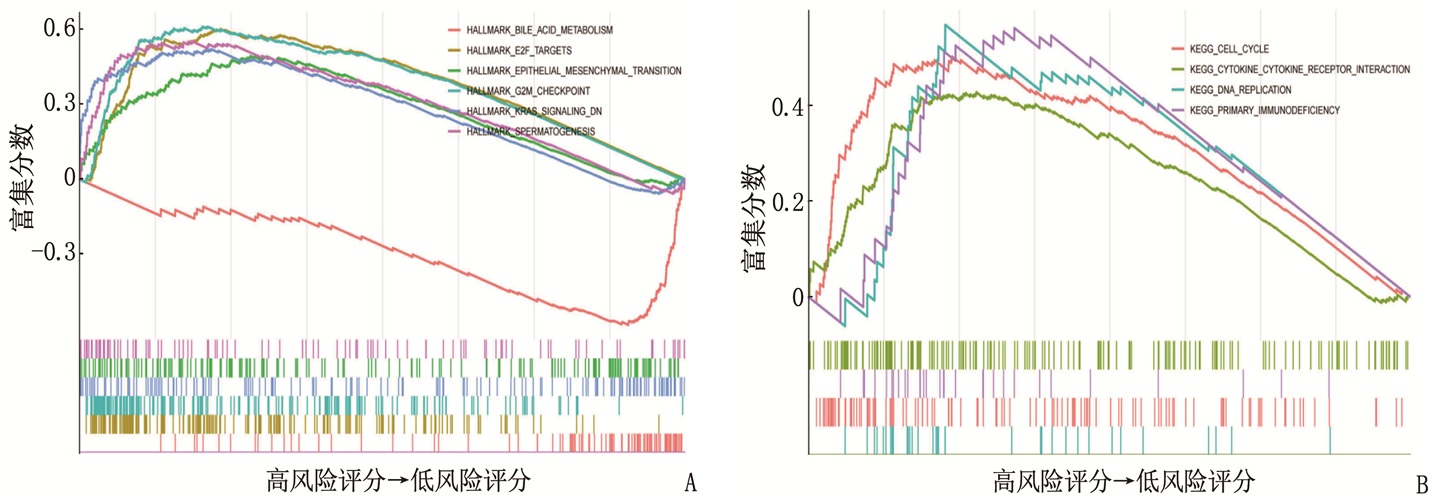
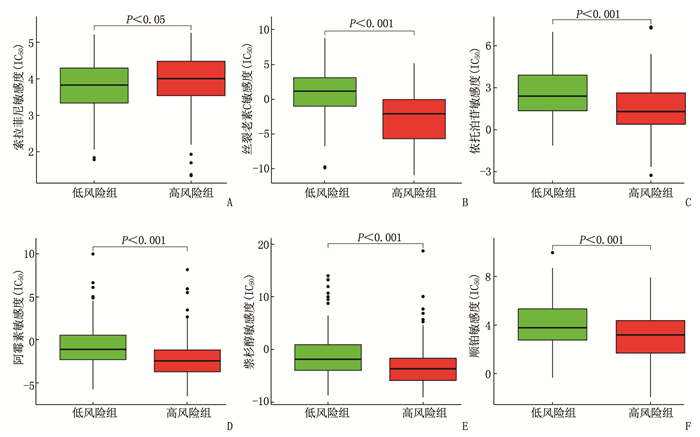
摘要:目的 利用公共数据库构建用于临床治疗肝细胞癌(HCC)的预后风险模型。方法 分别从癌症基因组图谱(TCGA)和国际癌症基因组联盟(ICGC)下载HCC以及癌旁正常组织的mRNA表达数据及临床信息。在TCGA队列中筛选与总生存期(OS)相关的差异表达基因(DEGs), 从中抽取2个或3个mRNAs构成一个组合, 对所有组合进行Cox风险比例回归模型构建。通过受试者工作特征(ROC)曲线的曲线下面积(AUC)确定最优基因组合,并进行基于ICGC队列的外部验证; 以TCGA队列的风险评分中位值将患者分为高风险组与低风险组,进行基因集富集分析(GSEA), 并通过pRRophetic R软件包预测HCC患者使用索拉非尼、丝裂霉素、依托泊苷、阿霉素、紫杉醇和顺铂的相对半抑制浓度(IC50)。结果 该预后风险模型预测TCGA队列的1、3、5年OS的ROC的AUC分别是0.786、0.713、0.699, 预测ICGC队列的1、3、4年OS的ROC的AUC分别为0.719、0.709、0.766。GSEA表明高风险组患者细胞周期相关通路被激活,胆汁酸代谢被抑制。索拉非尼在低风险组的IC50低于高风险组,而细胞周期相关化疗药物在低风险组的IC50高于高风险组,差异均有统计学意义(P < 0.05)。结论 本研究建立并验证了HCC预后风险模型,为HCC患者个体化诊疗方案的制订提供参考依据。Abstract:Objective To construct a prognostic risk model for clinical treatment of hepatocellular carcinoma (HCC) based on public databases.Methods The mRNA expression data and clinical information of HCC and adjacent normal tissues were downloaded from The Cancer Genome Atlas (TCGA) and International Cancer Genome Consortium (ICGC). Differentially expressed genes (DEGs) related to overall survival (OS) were screened in the TCGA cohort, 2 or 3 mRNAs were selected to form a combination, and Cox risk proportional regression model was constructed for all combinations. The optimal gene combination was determined by the area under the curve (AUC) of the receiver operating characteristic (ROC) curve, and external validation based on ICGC cohort was carried out. The patients were divided into high-risk group and low-risk group according to the median risk score of TCGA cohort, gene set enrichment analysis (GSEA) was performed, and the relative half-inhibitory concentrations (IC50) of sorafenib, mitomycin, etoposide, adriamycin, paclitaxel and cisplatin in HCC patients were predicted by pRRophetic R software package.Results For this prognostic risk model, the AUC of the ROC curve for predicting 1-, 3- and 5-year OS in the TCGA cohort were 0.786, 0.713 and 0.699, respectively, and the AUC of the ROC curve for predicting 1-, 3- and 4-year OS in the ICGC cohort were 0.719, 0.709 and 0.766, respectively. GSEA revealed that cell cycle related pathways were activated and bile acid metabolism was inhibited in the high-risk group. The IC50 of sorafenib in the low-risk group was significantly lower than that in the high-risk group, while the IC50 of cell cycle related chemotherapy drugs in the low-risk group was significantly higher than that in the high-risk group (P < 0.05).Conclusion This study establishes and verifies the prognostic risk model for HCC, and provides a reference for the formulation of individualized diagnosis and treatment plan for HCC patients.
-
肺癌目前是中国发病率、死亡率最高的恶性肿瘤, 5年生存率低于18%[1]。非小细胞肺癌(NSCLC)在肺癌中占比为85%, 其治疗方式主要有手术、化放疗、靶向药物治疗等[2]。研究[3]发现,多数NSCLC患者存在表皮生长因子受体(EGFR)基因突变,且EGFR合并肿瘤抑制基因共同突变的患者往往预后不良。EGFR酪氨酸激酶抑制剂(EGFR-TKI)是EGFR突变NSCLC患者的首选治疗方案,但治疗一段时间后会出现耐药现象,影响预后[4]。
微小RNA(miRNA)是广泛参与机体各种生命活动的单链小分子非编码RNA, 其在肿瘤中的差异表达及在肿瘤细胞增殖、凋亡、侵袭、化疗抵抗、耐药等方面的调控作用已被广泛报道[5-7]。研究[8-9]发现, miR-338-3p在肺癌等多种肿瘤中表达下调,有望成为NSCLC治疗的潜在靶点。研究[10]显示, miR-338-3p可通过EGFR信号通路抑制NSCLC细胞增殖、迁移及侵袭,但对NSCLC细胞出现EGFR-TKI耐药的作用研究较少。本研究探讨miR-338-3p对EGFR-TKI吉非替尼耐药的肺癌细胞株PC-9/GR凋亡及程序性死亡配体1(pPD-L1)表达的影响,并初步分析其作用机制。
1. 材料与方法
1.1 试剂与仪器
人肺腺癌细胞株PC-9及其吉非替尼耐药细胞株PC-9/GR(货号YS2301C、YS3629C)购自美国ATCC; RPMI-1640培养液(货号CP680110A)购自上海冠导生物工程有限公司; 吉非替尼、Annexin V-FITC/PI凋亡检测试剂盒、信号转导和转录激活因子1(STAT1)抑制剂Fludarabine(货号JKLN007404a、JKMK1417B、JKLN020840)购自上海经科化学科技有限公司; MTT溶液(货号JK3297)购自上海晶抗生物工程有限公司; miR-338-3p NC、miR-338-3p模拟物及miR-338-3p、U6引物购自柏业贸易(上海)有限公司; 兔抗p-STAT1、兔抗STAT1、兔抗PD-L1、兔抗Bcl-2、兔抗Bax、兔抗GAPDH、羊抗兔二抗(货号ab109461、ab210524、ab243877、ab196495、ab232479、ab181603、ab133470)购自美国abcam。NAPCO-8800型培养箱购自美国Shellab; MODEL550型酶标仪、GS-800型凝胶扫描成像系统购自美国Bio-Rad; Lightcycler 480II型荧光定量PCR仪购自德国Roche; CytoFLEX型流式细胞仪购自美国Beckman coulter。
1.2 方法
1.2.1 细胞培养与分组
PC-9细胞、PC-9/GR细胞复苏后接种于含10%胎牛血清的RPMI-1640培养液,放入37 ℃、5% CO2细胞培养箱中,生长至覆盖培养瓶80%面积,弃培养液,用0.25%胰蛋白酶消化,显微镜下观察到细胞回缩变圆后终止消化,吹打为单细胞并更换新鲜培养液重悬,按1∶2比例分瓶传代。
取传代3次的对数生长期PC-9、PC-9/GR细胞,按2×104个/mL接种于细胞培养板,分别加0、0.25、0.5、1、2、4、8 μmol/L吉非替尼,检测细胞增殖情况,以确定吉非替尼用量。此外,传代3次的对数生长期PC-9、PC-9/GR细胞按2×104个/mL接种于细胞培养板培养后,检测细胞中miR-338-3p表达。
将PC-9/GR细胞分为对照组、miR-338-3p NC组、miR-338-3p组和STAT1抑制剂组。其中miR-338-3p NC组、miR-338-3p组、STAT1抑制剂组细胞贴壁后,分别转染miR-338-3p NC、miR-338-3p模拟物和miR-338-3p模拟物,培养48 h后采用实时荧光定量聚合酶链反应(qRT-PCR)检测转染效果,之后4组均在培养液中加1 μmol/L吉非替尼, STAT1抑制剂组同时加100 μmol/L的STAT1抑制剂Fludarabine,细胞继续培养,进行后续实验。
1.2.2 qRT-PCR检测细胞中miR-338-3p表达
细胞培养48 h, 使用TRIzol提取细胞总RNA并检测其浓度、纯度,以RNA为模板行逆转录合成cDNA后进行qRT-PCR反应,用荧光定量PCR仪检测,U6为内参基因, miR-338-3p表达结果以2-△△Ct法表示。miR-338-3p上游引物: 5′-GTCAGTTCCAGCATCAGTGATT-3′, 下游引物: 5′-GTGCAGGGTCCGAGGT-3′; U6上游引物: 5′-GCTCGCTTCGGCAGCACA-3′, 下游引物: 5′-GAGGTATTCGCA CCAGAGGA-3′。
1.2.3 MTT法检测细胞增殖
细胞培养48 h, 每孔加10 μL MTT溶液,培养4 h, 弃上清,每孔加150 μL DMSO, 震荡5 min, 用酶标仪检测490 nm波长处吸光度(OD)值,计算增殖率(%)=OD实验组/OD对照组×100%。
1.2.4 流式细胞仪检测细胞凋亡
细胞培养48 h, 收集后按照Annexin V-FITC/PI凋亡试剂盒说明书进行处理,避光孵育15 min, 使用流式细胞仪进行检测。
1.2.5 蛋白印迹(WB)法检测细胞中p-STAT1、STAT1、PD-L1、Bcl-2、Bax蛋白表达情况
细胞培养48 h, 收集细胞,使用RIPA裂解液提取总蛋白,BCA法定量。取20 μg蛋白样本, 95 ℃水浴5 min使蛋白变性,上样,十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)分离蛋白,转移目的蛋白至聚偏二氟乙烯(PVDF)膜,用封闭液封闭1.5 h, 放入一抗(兔抗p-STAT1、兔抗STAT1、兔抗PD-L1、兔抗Bcl-2、兔抗Bax、兔抗GAPDH)稀释液(均1∶1 500)中孵育过夜,放入羊抗兔二抗(辣根过氧化物酶标记)稀释液(1∶2 000)中室温孵育1 h, 化学发光法显色,凝胶扫描成像系统扫描图像,分析条带灰度值。目的蛋白表达量以目的蛋白与GAPDH灰度值的比值表示。
1.3 统计学分析
采用SPSS 21.0软件进行数据分析。数据以(x±s)表示,多组间比较采用单因素方差分析,两两比较进行SNK-q检验。P < 0.05表示差异有统计学意义。
2. 结果
2.1 PC-9细胞、PC-9/GR细胞在不同浓度吉非替尼作用下的增殖率
随着吉非替尼浓度逐渐升高, PC-9细胞、PC-9/GR细胞的增殖率呈逐渐降低趋势(P < 0.05); 当吉非替尼浓度升高至1.00 μmol/L时, PC-9细胞与PC-9/GR细胞增殖率比较,差异有统计学意义(P < 0.05)。故后续实验均采用1.00 μmol/L作为吉非替尼的处理浓度。见表 1。
表 1 不同浓度吉非替尼作用下PC-9细胞、PC-9/GR细胞的增殖率比较(n=6)(x±s)吉非替尼浓度/(μmol/L) 增殖率/% PC-9细胞 PC-9/GR细胞 0 100.00±0 100.00±0 0.25 92.53±4.48* 94.12±4.79 0.50 82.74±4.16* 86.23±4.38* 1.00 70.80±4.05* 76.72±4.16*# 2.00 58.16±4.25* 65.02±4.14*# 4.00 49.89±4.50* 57.64±4.42*# 8.00 40.06±3.88* 49.22±3.95*# 与0 μmol/L比较, *P < 0.05; 与PC-9细胞比较, #P < 0.05。 2.2 PC-9细胞、PC-9/GR细胞中miR-338-3p表达情况
与PC-9细胞miR-338-3p表达水平(1.00±0)相比, PC-9/GR细胞中miR-338-3p表达水平(0.84±0.12)降低,差异有统计学意义(t=3.266, P=0.008)。
2.3 转染后PC-9/GR细胞miR-338-3p表达情况
对照组与miR-338-3p NC组的miR-338-3p表达水平比较,差异无统计学意义(P > 0.05); 与对照组、miR-338-3p NC组相比, miR-338-3p组、STAT1抑制剂组的miR-338-3p表达水平均升高,差异有统计学意义(P < 0.05); miR-338-3p组与STAT1抑制剂组的miR-338-3p表达水平比较,差异无统计学意义(P > 0.05)。见表 2。
表 2 转染后PC-9/GR细胞miR-338-3p表达水平比较(n=6)(x±s)组别 miR-338-3p 对照组 1.00±0 miR-338-3p NC组 1.12±0.15 miR-338-3p组 2.20±0.17*# STAT1抑制剂组 2.16±0.18*# 与对照组比较, *P < 0.05;
与miR-338-3p NC组比较, #P < 0.05。2.4 各组PC-9/GR细胞增殖情况
对照组与miR-338-3p NC组的PC-9/GR细胞增殖率比较,差异无统计学意义(P > 0.05); 与miR-338-3p NC组相比, miR-338-3p组PC-9/GR细胞增殖率降低,差异有统计学意义(P < 0.05); 与miR-338-3p组相比, STAT1抑制剂组PC-9/GR细胞增殖率升高,差异有统计学意义(P < 0.05)。见表 3。
表 3 各组PC-9/GR细胞增殖率比较(n=6)(x±s)组别 增殖率/% 对照组 100.00±0 miR-338-3p NC组 98.63±5.65 miR-338-3p组 52.35±5.02* STAT1抑制剂组 82.68±5.46# 与miR-338-3p NC组比较, *P < 0.05;
与miR-338-3p组比较, #P < 0.05。2.5 各组PC-9/GR细胞凋亡情况
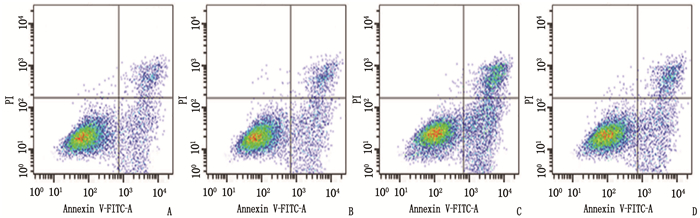
对照组与miR-338-3p NC组的PC-9/GR细胞凋亡率比较,差异无统计学意义(P > 0.05); 与miR-338-3p NC组相比, miR-338-3p组PC-9/GR细胞凋亡率升高,差异有统计学意义(P < 0.05); 与miR-338-3p组相比, STAT1抑制剂组PC-9/GR细胞凋亡率降低,差异有统计学意义(P < 0.05)。见图 1、表 4。
表 4 各组PC-9/GR细胞凋亡率比较(n=6)(x±s)组别 凋亡率/% 对照组 18.56±1.55 miR-338-3p NC组 18.04±1.60 miR-338-3p组 35.92±2.69* STAT1抑制剂组 22.83±2.15# 与miR-338-3p NC组比较, *P < 0.05;
与miR-338-3p组比较, #P < 0.05。2.6 各组PC-9/GR细胞中p-STAT1、STAT1、PD-L1、Bcl-2、Bax蛋白表达情况
对照组与miR-338-3p NC组的PC-9/GR细胞中p-STAT1/STAT1、PD-L1、Bcl-2、Bax蛋白表达水平比较,差异无统计学意义(P > 0.05); 与miR-338-3p NC组相比, miR-338-3p组PC-9/GR细胞中p-STAT1/STAT1、Bax蛋白表达水平升高, PD-L1、Bcl-2蛋白表达水平降低,差异有统计学意义(P < 0.05); 与miR-338-3p组相比, STAT1抑制剂组PC-9/GR细胞中p-STAT1/STAT1、Bax蛋白表达水平降低, PD-L1、Bcl-2蛋白水平升高,差异有统计学意义(P < 0.05)。见图 2、表 5。
表 5 各组PC-9/GR细胞中p-STAT1、STAT1、PD-L1、Bcl-2、Bax蛋白表达水平比较(n=6)(x±s)组别 p-STAT1/STAT1 PD-L1/GAPDH Bcl-2/GAPDH Bax/GAPDH 对照组 0.45±0.06 0.72±0.06 0.80±0.07 0.43±0.05 miR-338-3p NC组 0.47±0.07 0.71±0.07 0.83±0.07 0.40±0.05 miR-338-3p组 0.83±0.07* 0.39±0.05* 0.40±0.04* 0.88±0.06* STAT1抑制剂组 0.60±0.06# 0.58±0.06# 0.65±0.05# 0.57±0.06# 与miR-338-3p NC组比较, *P < 0.05; 与miR-338-3p组比较, #P < 0.05。 3. 讨论
肺癌是全球肿瘤相关死亡的主要原因,近年来其发病率不断升高。EGFR是原癌基因c-erb B1的表达产物,属于酪氨酸激酶受体,在多种肿瘤中高表达[11]。研究[12-13]发现,存在EGFR突变的亚洲肺癌患者占50%, 这部分人群体内肺癌细胞增殖、生长依赖于突变的EGFR, 用EGFR-TKI对其进行治疗,临床疗效较好,但治疗1年内均会出现获得性耐药。故解决EGFR-TKI耐药问题是肺癌治疗方面比较棘手的问题。吉非替尼是第一代EGFR-TKI, 本研究通过设置吉非替尼浓度梯度,检测PC-9细胞、PC-9/GR细胞的增殖率,最终选择1.00 μmol/L作为后续实验中PC-9/GR细胞的吉非替尼用药浓度。
研究[14-15]表明, miRNA在肿瘤组织中异常表达,发挥促癌或抑癌基因的作用,参与肿瘤发生及进展。由于miRNA具有肿瘤病理的特异性,常被认为可作为一种新型靶点用于肿瘤的靶向治疗。研究[16]显示, miRNA不仅可介导肿瘤细胞增殖、凋亡、迁移等生物学行为,在肿瘤发生放化疗抵抗、耐药等过程中亦发挥重要作用。miR-338-3p是miR-338家族成员,在食道癌、头颈部鳞状细胞癌、肾嫌色细胞癌、肾乳头状肾细胞癌、肺鳞癌及甲状腺癌中表达显著下调,而在肝细胞癌、结肠腺癌及肾透明细胞癌中表达显著上调[17-18]。本研究发现, PC-9/GR细胞中miR-338-3p表达水平较PC-9细胞显著降低,提示miR-338-3p在EGFR-TKI耐药的PC-9细胞中差异表达,其低表达可能参与PC-9细胞发生EGFR-TKI耐药的机制。通过转染miR-338-3p模拟物,上调miR-338-3p在PC-9/GR细胞中的表达水平后, PC-9/GR细胞增殖率显著降低,凋亡率显著升高,细胞中Bcl-2家族促凋亡成员Bax蛋白表达水平显著升高,抑凋亡成员Bcl-2蛋白水平[19]显著降低,提示上调miR-338-3p可一定程度逆转PC-9/GR细胞的EGFR-TKI耐药,抑制细胞增殖,并诱导细胞凋亡。PD-L1是在多种肿瘤细胞表面高表达的因子,可帮助肿瘤细胞发生免疫逃逸,其高表达往往与肿瘤预后不良呈正相关[20-22]。上调miR-338-3p表达后, PC-9/GR细胞中PD-L1表达水平显著降低,提示miR-338-3p可能抑制PC-9/GR细胞发生免疫逃逸,进而促进细胞凋亡。
STAT1是一种信号传导与转录活化因子蛋白,能从各个角度参与对细胞生长、分化、存活及凋亡的调控,目前多被认为是肿瘤抑制因子[23-24]。孙思博等[25]研究表明, STAT家族成员在NSCLC获得性耐药过程中发挥重要作用。本研究显示, miR-338-3p表达上调后, PC-9/GR细胞中p-STAT1/STAT1蛋白表达水平显著升高,提示STAT1激活参与miR-338-3p逆转PC-9/GR细胞的EGFR-TKI耐药过程。在miR-338-3p表达上调基础上,加入STAT1抑制剂抑制STAT1激活后, miR-338-3p表达无显著变化,细胞增殖率显著升高,凋亡率显著降低,细胞中Bax蛋白表达水平显著降低, PD-L1、Bcl-2蛋白表达水平显著升高,进一步提示miR-338-3p逆转PC-9/GR细胞的EGFR-TKI耐药过程可能与激活STAT1有关。
综上所述, miR-338-3p表达上调可激活STAT1, 抑制EGFR-TKI耐药肺癌细胞株PC-9/GR细胞中PD-L1表达,并诱导细胞凋亡,这可能为临床解决EGFR-TKI耐药问题提供理论依据。
-
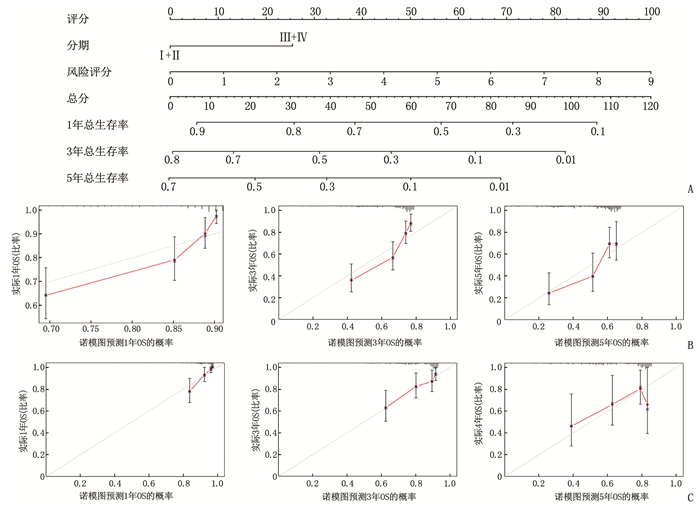
表 1 HCC患者的临床病理资料[n(%)]
临床资料 TCGA队列
(n=365)ICGC队列
(n=231)年龄/岁 61(16, 90) 69(31, 89) 性别 女 119(32.6) 61(26.4) 男 246(67.4) 170(72.6) 肿瘤分级 Ⅰ级 55(15.1) — Ⅱ级 175(47.9) — Ⅲ级 118(32.3) — Ⅳ级 12(3.3) — 未知 5(1.4) — 肿瘤分期 Ⅰ期 170(46.6) 36(15.6) Ⅱ期 84(23.0) 105(45.5) Ⅲ期 83(22.7) 71(30.7) Ⅳ期 4(1.1) 19(8.2) 未知 24(6.6) 0 血管浸润 有 106(29.0) — 无 205(56.2) — 未知 54(14.8) — 甲胎蛋白 ≤200 ng/mL 201(55.1) — > 200 ng/mL 75(20.5) — 未知 89(24.4) — 年龄以中位数(最小值,最大值)表示。 表 2 风险评分与HCC患者临床病理资料的相关性
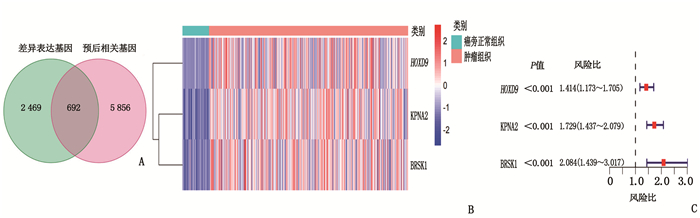
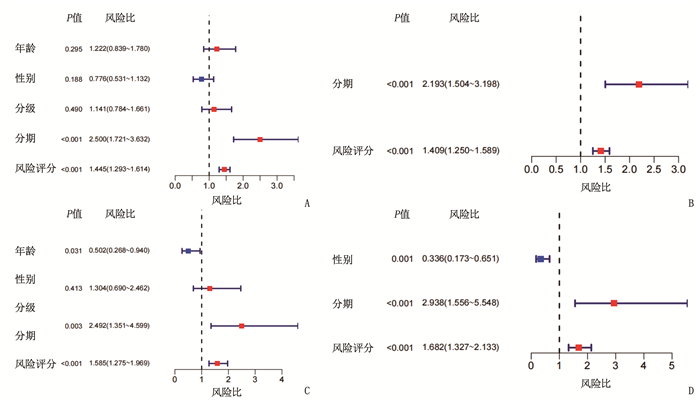
临床资料 TCGA队列 ICGC队列 高风险组 低风险组 P 高风险组 低风险组 P 性别 0.80 1.00 女性 58 61 22 39 男性 125 121 62 108 年龄 0.80 0.42 < 60岁 81 84 29 60 ≥60岁 102 98 55 87 肿瘤分级 < 0.05 - 1、2级 134 96 - - 3、4级 47 83 - - 肿瘤分期 < 0.05 < 0.05 Ⅰ、Ⅱ期 141 113 63 78 Ⅲ、Ⅳ期 30 57 21 69 血管浸润 < 0.05 - 无 124 81 - - 有 44 62 - - 甲胎蛋白 < 0.05 - ≤200 ng/mL 122 79 - - >200 ng/mL 27 48 - - -
[1] VILLANUEVA A. Hepatocellular carcinoma[J]. N Engl J Med, 2019, 380(15): 1450-1462. doi: 10.1056/NEJMra1713263
[2] YANG J D, HAINAUT P, GORES G J, et al. A global view of hepatocellular carcinoma: trends, risk, prevention and management[J]. Nat Rev Gastroenterol Hepatol, 2019, 16(10): 589-604. doi: 10.1038/s41575-019-0186-y
[3] TORRE L A, BRAY F, SIEGEL R L, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87-108. doi: 10.3322/caac.21262
[4] BROWN Z J, GRETEN T F, HEINRICH B. Adjuvant treatment of hepatocellular carcinoma: prospect of immunotherapy[J]. Hepatology, 2019, 70(4): 1437-1442. doi: 10.1002/hep.30633
[5] HUANG W T, SKANDERUP A J, LEE C G. Advances in genomic hepatocellular carcinoma research[J]. Gigascience, 2018, 7(11): 1-22. http://www.onacademic.com/detail/journal_1000041617495499_6d02.html
[6] DOMINGUEZ D A, WANG X W. Impact of next-generation sequencing on outcomes in hepatocellular carcinoma: how precise are we really[J]. J Hepatocell Carcinoma, 2020, 7: 33-37. doi: 10.2147/JHC.S217948
[7] CARUSO S, O'BRIEN D R, CLEARY S P, et al. Genetics of hepatocellular carcinoma: approaches to explore molecular diversity[J]. Hepatology, 2021, 73(Suppl 1): 14-26. http://www.researchgate.net/publication/348322949_Genetics_of_Hepatocellular_Carcinoma_Approaches_to_Explore_Molecular_Diversity
[8] GEELEHER P, COX N, HUANG R S. pRRophetic: an R package for prediction of clinical chemotherapeutic response from tumor gene expression levels[J]. PLoS One, 2014, 9(9): e107468. doi: 10.1371/journal.pone.0107468
[9] PAN Y S, CHEN H R, YU J. Biomarkers in hepatocellular carcinoma: current status and future perspectives[J]. Biomedicines, 2020, 8(12): 576. doi: 10.3390/biomedicines8120576
[10] OURA K, MORISHITA A, MASAKI T. Molecular and functional roles of microRNAs in the progression of hepatocellular carcinoma-A review[J]. Int J Mol Sci, 2020, 21(21): 8362. doi: 10.3390/ijms21218362
[11] KIM E, VIATOUR P. Hepatocellular carcinoma: old friends and new tricks[J]. Exp Mol Med, 2020, 52(12): 1898-1907. doi: 10.1038/s12276-020-00527-1
[12] MUINAO T, DEKA BORUAH H P, PAL M. Multi-biomarker panel signature as the key to diagnosis of ovarian cancer[J]. Heliyon, 2019, 5(12): e02826. doi: 10.1016/j.heliyon.2019.e02826
[13] FOUNTZILAS C, KAKLAMANI V G. Multi-gene panel testing in breast cancer management[J]. Cancer Treat Res, 2018, 173: 121-140.
[14] YANG Z C, ZI Q, XU K, et al. Development of a macrophages-related 4-gene signature and nomogram for the overall survival prediction of hepatocellular carcinoma based on WGCNA and LASSO algorithm[J]. Int Immunopharmacol, 2021, 90: 107238. doi: 10.1016/j.intimp.2020.107238
[15] LIU G M, XIE W X, ZHANG C Y, et al. Identification of a four-gene metabolic signature predicting overall survival for hepatocellular carcinoma[J]. J Cell Physiol, 2020, 235(2): 1624-1636. doi: 10.1002/jcp.29081
[16] LIN P, HE R Q, DANG Y W, et al. An autophagy-related gene expression signature for survival prediction in multiple cohorts of hepatocellular carcinoma patients[J]. Oncotarget, 2018, 9(25): 17368-17395. doi: 10.18632/oncotarget.24089
[17] LI G X, XU W Q, ZHANG L, et al. Development and validation of a CIMP-associated prognostic model for hepatocellular carcinoma[J]. EBioMedicine, 2019, 47: 128-141. doi: 10.1016/j.ebiom.2019.08.064
[18] YU J, WU X L, LV M, et al. A model for predicting prognosis in patients with esophageal squamous cell carcinoma based on joint representation learning[J]. Oncol Lett, 2020, 20(6): 387. http://www.ncbi.nlm.nih.gov/pubmed/33193847
[19] YANG Y J, WANG C Y, WEI N D, et al. Identification of prognostic chromatin-remodeling genes in clear cell renal cell carcinoma[J]. Aging (Albany NY), 2020, 12(24): 25614-25642.
[20] HONG W F, LIANG L, GU Y J, et al. Immune-related lncRNA to construct novel signature and predict the immune landscape of human hepatocellular carcinoma[J]. Mol Ther Nucleic Acids, 2020, 22: 937-947. doi: 10.1016/j.omtn.2020.10.002
[21] 陈懿, 李雪, 林文雅, 等. 自噬基因预测肝癌患者长期生存及通路分析[J]. 医学研究杂志, 2021, 50(1): 137-141. https://www.cnki.com.cn/Article/CJFDTOTAL-YXYZ202101031.htm [22] 段万里, 任伟, 邓骞, 等. 基于TCGA数据库的肾癌自噬相关基因预后模型的建立与应用[J]. 现代泌尿外科杂志, 2020, 25(10): 870-875, 889. doi: 10.3969/j.issn.1009-8291.2020.10.003 [23] TABUSE M, OHTA S, OHASHI Y, et al. Functional analysis of HOXD9 in human gliomas and glioma cancer stem cells[J]. Mol Cancer, 2011, 10: 60. doi: 10.1186/1476-4598-10-60
[24] LONG J Y, ZHANG L, WAN X S, et al. A four-gene-based prognostic model predicts overall survival in patients with hepatocellular carcinoma[J]. J Cell Mol Med, 2018, 22(12): 5928-5938. doi: 10.1111/jcmm.13863
[25] LV X P, LI L L, LV L, et al. HOXD9 promotes epithelial-mesenchymal transition and cancer metastasis by ZEB1 regulation in hepatocellular carcinoma[J]. J Exp Clin Cancer Res, 2015, 34: 133. doi: 10.1186/s13046-015-0245-3
[26] CHRISTIANSEN A, DYRSKJOT L. The functional role of the novel biomarker karyopherin α 2 (KPNA2) in cancer[J]. Cancer Lett, 2013, 331(1): 18-23. doi: 10.1016/j.canlet.2012.12.013
[27] GUO X G, WANG Z H, ZHANG J N, et al. Upregulated KPNA2 promotes hepatocellular carcinoma progression and indicates prognostic significance across human cancer types[J]. Acta Biochim Biophys Sin (Shanghai), 2019, 51(3): 285-292. doi: 10.1093/abbs/gmz003
[28] JIANG P, TANG Y Q, HE L, et al. Aberrant expression of nuclear KPNA2 is correlated with early recurrence and poor prognosis in patients with small hepatocellular carcinoma after hepatectomy[J]. Med Oncol, 2014, 31(8): 1-7.
[29] KONIROVA J, OLTOVA J, CORLETT A, et al. Modulated DISP3/PTCHD2 expression influences neural stem cell fate decisions[J]. Sci Rep, 2017, 7: 41597. doi: 10.1038/srep41597
[30] XIE G X, WANG X N, HUANG F J, et al. Dysregulated hepatic bile acids collaboratively promote liver carcinogenesis[J]. Int J Cancer, 2016, 139(8): 1764-1775. doi: 10.1002/ijc.30219
[31] CHEN W B, OU M L, TANG D E, et al. Identification and validation of immune-related gene prognostic signature for hepatocellular carcinoma[J]. J Immunol Res, 2020, 2020: 5494858. http://qikan.cqvip.com/Qikan/Article/Detail?id=7106408960
[32] STACEY D, KAZLAUSKAS A. Regulation of Ras signaling by the cell cycle[J]. Curr Opin Genet Dev, 2002, 12(1): 44-46. doi: 10.1016/S0959-437X(01)00262-3
[33] MATSUDA Y. Molecular mechanism underlying the functional loss of cyclindependent kinase inhibitors p16 and p27 in hepatocellular carcinoma[J]. World J Gastroenterol, 2008, 14(11): 1734-1740. doi: 10.3748/wjg.14.1734
[34] MATSUDA Y, ICHIDA T. p16 and p27 are functionally correlated during the progress of hepatocarcinogenesis[J]. Med Mol Morphol, 2006, 39(4): 169-175. doi: 10.1007/s00795-006-0339-2
[35] GREENBAUM L E. Cell cycle regulation and hepatocarcinogenesis[J]. Cancer Biol Ther, 2004, 3(12): 1200-1207. doi: 10.4161/cbt.3.12.1392
-
期刊类型引用(4)
1. 刘雪,张静智,曾玲. 橡皮圈联合金属夹的内牵引技术在内镜黏膜下剥离术中的临床应用. 中国当代医药. 2024(02): 37-40 .  百度学术
百度学术
2. 汪茂莎,王倩,李雯,陈婷,王静,刘风,张颖. 消化内镜护士人体工效学负荷评价量表的研制与评价. 实用临床医药杂志. 2024(12): 138-143+148 .  本站查看
本站查看
3. 陈佳,侯向红,石铭鸿,杨洁,姜瑞. 早期结直肠病变患者行内镜下黏膜剥离术后发生电凝综合征的高危因素. 中外医学研究. 2023(23): 147-150 .  百度学术
百度学术
4. 陈佳,侯向红,石铭鸿,杨洁,姜瑞. 预切开内镜下黏膜切除术与内镜黏膜下剥离术在结直肠侧向发育型肿瘤患者中应用对比. 中国医学创新. 2023(27): 5-8 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载:

 苏公网安备 32100302010246号
苏公网安备 32100302010246号

